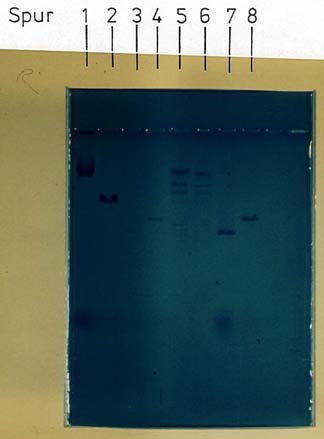
Bei der Bande im Bereich von 2 kb der Spuren 1, 2, 4 und 5 der Abbildung 5 handelt es sich um die superhelikale Form des Plasmids pUC 19. Diesen Schluß läßt zumindest der Vergleich mit Spur 6 zu, in der eine DNA-Probe des Plasmids beigegeben wurde. Dort befindet sich ebenfalls eine Bande im Bereich von 2 kb. Die nachfolgenden Banden in Spur 1 sind mit der entspannten und der linearisierten Konformation in Einklang zu bringen. Ob es sich bei der Bande in 23 kb Bereich der Spur 1 um ein Multimer des Plasmids oder um chromosomale Bakterien-DNA handelt, ist nicht einwandfrei zu entscheiden. Das Bandenmuster aus Spur 1 tritt sonst in keiner der anderen Spuren auf. Deshalb wurde wegen der Vergleichbarkeit der Ergebnisse nur die superhelikale Form des Plasmids für die Errechnung isolierter DNA-Mengen herangezogen (Tab. 7).
Grundsätzlich war es mit beiden Methoden möglich, Plasmide zu isolieren
(Tab. 8, Tab. 9, Tab. 10 und Tab.11). Hinsichtlich der Plasmidausbeute konnten unter
vergleichbaren Voraussetzungen (gleiche Übernachtkulturen, gleiche Anfangsvolumina von
Bakterien, gleiche Zentrifugationszeiten) mittels alkalischer Lyse bis auf eine Ausnahme
höhere Plasmiderträge erzielt werden als durch die Methode der Kochlyse (Tab. 9). Dabei
lagen die Erträge bei aus Flüssigübernachtkulturen gewonnener Plasmid-DNA
grundsätzlich höher als bei der Plasmidpräparation aus direkt von Agarnährböden
gepickten Bakterien (Tab. 8).
Berücksichtigt man die übrigen Resultate (Tab. 10 und 11), so zeigen sich vor allem
bei den aus einer für 12 - 24 Stunden bei 37 °C inkubierten Flüssigkultur erzielten
Ergebnissen an isolierter Plasmid-DNA relativ starke Schwankungen. Bei insgesamt 26
Plasmidpräparationen, wobei 13 nach der Methode der alkalischen Lyse und 11 nach der
Methode der Kochlyse durchgeführt wurden, konnte eine maximale Plasmidkonzentration von
bis zu 80 ng/µl bei der alkalischen Lyse und
72 ng/µl im Zuge der Kochlyse nachgewiesen werden. Es konnten
Plasmidkonzentrationen zwischen 1 ng/µl und 80 ng/µl dokumentiert werden. Bei fünf Präparationen wurde keine
Plasmid-DNA nachgewiesen. In allen fünf Fällen sollte das Plasmid pUC 18 isoliert werden.
Wesentliche Unterschiede zwischen der Präparationsmethode der alkalischen Lyse und der Kochlyse aus direkt von Agarnährböden gepickten Bakterienkolonien waren nicht festzustellen (Tab. 11).
Die erzielten Ergebnisse an isolierter Plasmid-DNA aus Bakterien, die direkt von einem
Agarnährboden gepickt wurden, waren durchweg geringer bzw. lagen unterhalb der mit
Ethidiumbromid nachzuweisenden DNA Menge von ca. 10 ng. Der Vergleich erfolgte mit den
Ergebnissen einer Plasmidisolation aus einer für 12 bis 24 h bei 37 °C inkubierten
Flüssigkultur (Tab. 10 und 11). Die Ursache für die schlechtere Ausbeute bei der
Isolation von Plasmid-DNA aus direkt von Agarnährbodenplatten gepickten Kolonien liegt
vermutlich in einer geringeren Zellzahl zu Beginn der Präparation. Eine Empfehlung
hinsichtlich der Anzahl der zu resuspendierenden Bakterienkolonien kann anhand der
Ergebnisse nicht eindeutig getroffen werden, da dieses auch von der Größe der Kolonien
abhängt. Als grobes Richtmaß sind pro Plasmidpräparation in etwa 10 Bakterienkolonien
mit einem ungefähren Durchmesser von 3 mm zu resuspendieren.
Allerdings ist die Methode der Plasmidpräparation aus direkt von Agarnährbodenplatten
gepickten Kolonien aufgrund der in der Regel geringen Menge an gewonnener Plasmid-DNA und
der Häufigkeit der Präparationen, bei denen keine Plasmid-DNA nachgewiesen werden
konnte, für Schulen nur bedingt zu empfehlen. Das gilt gerade auch vor dem Hintergrund
der Verwendung von Methylenblau als Färbemittel für DNA im Schülerexperiment zur
Gelelektrophorese, da die Nachweisgrenze für DNA in mit Methylenblau angefärbten Gelen ³ 40 ng beträgt. Deshalb sollte die Methode nur zusätzlich neben
einer Plasmidpräparation aus einer Flüssigkultur Verwendung finden.
Fehlende Lysozymzugabe wirkt sich ertragsmindernd aus (Tab. 12). Deshalb sollte man auf die Lysozymzugabe nicht verzichten, um über genügend Plasmid-DNA für die gelelektrophoretische Auftrennung mit anschließender Methylenblaufärbung und das Transformationsexperiment zu verfügen.
Ein ausgelassener Reinigungsschritt mit Ethanol am Ende der alkalischen Lyse wirkt sich
nicht negativ auf das Versuchsergebnis der Plasmidpräparation aus
(Tab. 13), so daß dieser Schritt im Versuchsprotokoll des Schülerexperimentes nicht
zwingend notwendig ist. Das gleiche kann für den Einsatz von RNase gesagt werden. Ohne
RNase-Zugabe konnte noch Plasmid-DNA in ausreichender Menge für die nachfolgenden
Teilexperimente gewonnen werden (Tab. 14). Die Plasmidpräparationen ohne Ethanolreinigung
und ohne RNase-Zugabe wurden auch für Transformationen verwendet, wobei gute Resultate
erzielt werden konnten
(Tab. 15). RNase wäre unter dem Gesichtspunkt eines „saubereren" Ergebnisses
bei der gelelektrophoretischen Auftrennung zu empfehlen, da dadurch die im Reaktionsansatz
befindliche RNA abgebaut und so das Auftreten einer Bande im niedermolekularen Bereich
vermieden wird.
Aufgrund ausreichender Mengen an isolierter Plasmid-DNA konnten für das Schülerteilexperiment der Plasmidpräparation nach alkalischer Methode die Zentrifugationszeiten von anfänglich 25 Minuten nach den Versuchsschritten, bei denen dem Reaktionsansatz Kaliumacetat zugeführt wird und die Isopropanolfällung stattfindet, auf 15 Minuten verkürzt werden. Eine weitere wesentliche Verkürzung der Zeiten kann wegen der dabei erzielten Mengen an gewonnener Plasmid-DNA nicht befürwortet werden (Tab. 16).
Die Angleichung der Bakterienmengen, aus denen Plasmide zu isolieren sind, erbrachte für beide Methoden ausreichende Versuchsergebnisse. Dabei wurde die Milliliterzahl bei der Kochlyse von 1,5 auf 3,0 ml zu Beginn der Plasmidpräparation erhöht, was letztendlich bei einer Verwendung von Reaktionsgefäßen mit einem Volumen von 1,5 ml einen zusätzlichen Zentrifugationsschritt bedeutet, der aber durch bessere Resultate gerechtfertigt ist. Bei der alkalischen Lyse wurde die anfängliche Milliliterzahl der Bakterien, aus denen Plasmide isoliert werden sollten, von 4,0 auf 3,0 ml gesenkt. Die dabei erzielten Resultate sind für eine gelelektrophoretische Auftrennung, die Transformation und ggf. eine Restriktionsspaltung ausreichend (Tab. 10).
Abschließend läßt sich sagen, daß nach den vorliegenden Versuchsprotokollen sowohl nach der Methode der alkalischen Lyse als auch nach der Kochlyse aus Flüssigkulturen Plasmide in ausreichender Menge gewonnen werden können. Aufgrund des Materialaufwandes und der benötigten Chemikalien kann keine der beiden Methoden bevorzugt für das Schülerexperiment empfohlen werden. Der Zeitaufwand für die Durchführung der Präparationen ist soweit optimiert worden, daß die Versuchsdurchführung nach beiden Methoden jeweils grundsätzlich in einer unterrichtlichen Doppelstunde (90 Minuten) möglich ist. Welche der beiden Methoden letztendlich in der Schule zum Einsatz kommt, sollte sich in erster Linie nach der schulischen Ausstattung mit den einzelnen Materialien und Chemikalien und den Möglichkeiten der Anschaffung richten.
Bei durchgeführten Restriktionsversuchen sollte überprüft werden, ob selbst isolierte Plasmid-DNA mit Restriktionsenzymen geschnitten werden kann. Dabei wurden sowohl Enzyme kommerzieller Anbieter als auch die aus dem sogenannten Lambda-Kit stammenden Enzyme getestet. Beim Lambda-Kit handelt es sich um ein Versuchskit für Schulen, der vom National Center for Biotechnology Education (NCBE) an der Universität Reading (England) in Kooperation mit dem Institut für die Pädagogik der Naturwissenschaften (IPN) in Kiel entwickelt wurde. Dabei sollen Schüler die im Kit mitgelieferte und getrocknete DNA des Bakteriophagen Lambda resuspendieren, mit Hilfe von ebenfalls in getrockneter Form vorliegenden Restriktionsnenzymen schneiden und anschließend elektrophoretisch im Agarosegel auftrennen. Alle dafür benötigten Materialien, bis auf die als Spannungsgeber dienenden Batterien (9V Blockbatterien), sind im Kit enthalten und können bei Bedarf einzeln beim NCBE nachbestellt werden. Die für die Restriktion verwendeten Enzyme (EcoRI, BamHI, HindIII) befinden sich bereits in separaten Reaktionsgefäßen, die zudem den jeweils spezifischen Puffer enthalten und somit direkt verwendet werden können. Desweiteren können die im Kit enthaltenen Enzyme bei Raumtemperatur gelagert werden.
Das für den Versuch verwendete Material und die angewandten Methoden sind bis auf die verwendeten Utensilien des Lambda-Kits mit den im Lehrermanual für das Schülerexperiment zur Restriktionsspaltung angegebenem Material und Methoden identisch (Kapitel 5.2.1). Die anschließende gelelektrophoretische Auftrennung erfolgte nach den Angaben im Lehrermanual zum Schülerexperiment Gelelektrophorese ( Kapitel 5.3.1).
Ergebnisse zur Restriktion von Plasmid-DNA mit nachfolgender gelelektrophoretischer
Auftrennung sind der Abb. 6 und Abb. 9 (S. 114) zu entnehmen.
Die Tabelle 17 zeigt die Effizienz von zwei getesteten Restriktionsenzymen, mit denen
unterschiedliche Plasmid-DNA geschnitten wurde.
|
Ansatz |
Enzym |
Herkunft |
Einheiten |
Plasmid |
Plasmidmenge |
Ergebnis |
|
1 |
EcoRI |
Lambda-Kit |
10 |
pBR 322 |
180 |
vollständige Spaltung |
|
2 |
BamHI |
Lambda-Kit |
10 |
pBR 322 |
180 |
vollständige Spaltung |
|
3 |
EcoRI |
Lambda-Kit |
10 |
pUC 18 |
160 |
vollständige Spaltung |
|
4 |
EcoRI |
Lambda-Kit |
10 |
pUC 18 |
100 |
vollständige Spaltung |
|
5 |
EcoRI |
Lambda-Kit |
10 |
pUC 19 |
300 |
vollständige Spaltung |
|
6 |
BamHI |
Lambda-Kit |
10 |
pUC 18 |
160 |
vollständige Spaltung |
|
7 |
BamHI |
Lambda-Kit |
10 |
pUC 18 |
160 |
vollständige Spaltung |
|
8 |
BamHI |
Lambda-Kit |
10 |
pUC 18 |
80 |
vollständige Spaltung |
|
9 |
EcoRI |
Boehringer |
4 |
pBR 322 |
300 |
vollständige Spaltung |
|
10 |
EcoRI |
Boehringer |
4 |
pUC 18 |
100 |
vollständige Spaltung |
|
11 |
EcoRI |
Boehringer |
4 |
pAmp |
180 |
vollständige Spaltung |
|
12 |
EcoRI |
Boehringer |
4 |
pUC 19 |
200 |
vollständige Spaltung |

| Als große Abbildung |
Alle getesteten Plasmide konnten mit den verwendeten Restriktionsenzymen geschnitten werden und zeigen ein durch ihre Linearisierung den Erwartungen entsprechendes verändertes Laufverhalten bei der Gelelektrophorese (Abb. 6). Die dabei durch Vergleich mit Marker-DNA ermittelten Plasmidgrößen in Kilobasen (kb) stimmen mit den in der Literatur angebenen Größen überein. Die jeweils eingesetzte Plasmidmenge konnte in allen Ansätzen vollständig geschnitten werden.
Die Durchführung eines solchen Experimentes im Rahmen des Schulunterrichtes sollte keine Schwierigkeiten bereiten. Bezogen auf die einzusetzende Menge von Restriktionsenzym ist zu fordern, daß eher zu viel als zu wenig Enzym dem Reaktionsansatz beigeben wird. Mit anderen Worten: Eine höhere Zugabe von Restriktionsenzymen (4 - 10 Einheiten) wirkt sich nicht negativ auf das Versuchsergebnis aus. Es kann vielmehr dadurch vermieden werden, daß die Spaltung unvollständig abläuft und mehr als nur eine erwartete DNA-Bande im Gel auftaucht. Ferner sind größere Enzymmengen bei einer Konzentration von 1 Einheit Enzym/ 0,125 µl auch erheblich besser zu pipettieren. Eine generelle Zugabe von 1 µl pro Restriktionsansatz ist unter den Gesichtspunkten zu empfehlen, vor allem auch wenn man bedenkt, daß 1 Einheit Enzym bei dem in den Bezugsquellen angegebenen Anbieter (Kapitel 3.3, Tab. 2) weniger als 0,01 DM kostet.
Falls Schulen den Lambda-Kit besitzen, können diese auf die dort enthaltenen Restriktionsenzyme zurückgreifen. Unter Umständen ist für Schulen die Anschaffung des Kits und die Verwendung von den darin enthaltenen Materialien kostengünstiger, als der Kauf der für den Laborbedarf bemessenden Mengen an Restriktionsenzymen, wo die kleinste abzugebende Menge 4000 Einheiten umfaßt und die Haltbarkeit der Enzyme begrenzt ist (nach Angaben der Hersteller bei voller Enzymaktivität 1 Jahr). Zudem können die Restriktionsenzyme des Lambda-Kits bei Raumtemperatur gelagert werden, wohingegen Restriktionsenzyme sonst in der Regel bei -20 °C aufbewahrt werden, wofür man entweder ein Dreisternekühlfach in einem Kühlschrank oder eine Tiefkühltruhe benötigt.
Die von kommerziellen Anbietern angebotenen Utensilien zur Elektrophorese sind nicht
billig und bei Preisen von ca. 2000,- DM für eine Elektrophoresekammer inkl. Kamm und
Netzgerät für den Schuletat kaum erschwinglich. Davon abweichend sollte bei
durchgeführten gelelektrophoretischen Analysen auf alternative Materialien
zurückgegriffen und auf ihre mögliche Verwendung im Schülerexperiment getestet werden.
Im Diskussionsteil werden aber auch Hinweise für den Bau einer Apparatur zur
Gelelektrophorese gegeben.
In einem zweiten Punkt sollten alternative Färbemethoden getestet werden, die anstelle
von Ethidiumbromid eingesetzt werden können.
Die Durchführung der Gelelektrophorese richtete sich nach den unter Kapitel 5.3.1
aufgeführten Hinweisen zur Gelelektrophorese.
Bei den getesteten alternativen Materialien handelt es sich zum einen um die aus dem
Lambda-Kit des NCBE und IPN stammenden Utensilien. Eine ausführliche Information über
die Verwendungsmöglichkeiten des Lambda-Kits findet sich im Kapitel 6.2.1. Die dort für
die Elektrophorese verwendeten Materialien sind für den Schulgebrauch zugeschnitten. Als
Gelkammer wird eine Kunststoffbox verwendet, die zum einen Ende hin eine Einkerbung für
den ebenfalls aus Kunststoff bestehenden Kamm besitzt. Der Kamm dient der Ausformung der
Taschen im Agarosegel. Die Spannungsquelle besteht hier aus ein oder zwei 9V
Blockbatterien, die in Reihe geschaltet werden können. Verbunden werden die Batterien mit
der Gelkammer über Krokodilklemmen, die an in die Kammer einzusetzende Kohlefasergewebe
angeschlossen werden, wodurch der Stromkreis geschlossen wird. Bei dem Kohlefasergewebe
handelt es sich um eine Entwicklung des NCBE und IPN (Abb. 7). Durch die Verwendung von
Batterien mit einer maximalen Spannung von 18V ist außerdem sichergestellt, daß die
Schüler vor schwerwiegenden oder tödlichen elektrischen Schlägen geschützt sind.
Als weitere alternative Spannungsquelle wurde die Verwendung eines
gleichstromliefernden Spielzeugeisenbahntransformators geprüft (Abb. 8).

|
| Abbildung 7: Die Materialien des Lambda-Kit für die Gelelektrophorese. |
 |
| Abbildung 8: Gleichstromliefernder Spielzeugeisenbahntransformator als alternative Spannungsquelle für die Gelelektrophorese. |
Bei den für die Anfärbung der DNA im Gel untersuchten alternativen Farbstoffen handelt es sich zum einen um das in einem unveröffentlichten Manuskript von NEVERS (o.J.) verwendete Methylenblau und den im Lambda-Kit verwendeten Farbstoff aus einer alkoholischen Lösung von Azur A.

| Als große Abbildung |
| Elektrodenmaterial | Stromfluß bei einer angelegten Spannung von | |
|
18 V |
9 V |
|
| V2A-Stahl |
5 mA |
2,5 mA |
| Kohlefasergewebe Lambda-Kit |
5 mA |
2,5 mA |
| Graphit (z.B. Bleistiftspitze) |
5 mA |
2,5 mA |
Wie bereits oben angesprochen sind die kommerziell angebotenen Materialien nicht sehr
billig und für den Schuletat kaum erschwinglich. Mit den im Lambda-Kit des NCBE und IPN
enthaltenen Materialien konnten gute Elektrophoreseergebnisse erzielt werden (Abb. 9), so
daß eine Verwendung im Schülerexperiment uneingeschränkt befürwortet werden kann.
Die in dem Kit verwendeten Materialien können zum größten Teil aber auch im Baumarkt
gekauft werden. Allerdings müssen diese noch z.T. für ihre Verwendung bearbeitet werden.
Zum Beispiel müßte der Kamm auf die entsprechende Kunststoffbox, die als Gelkammer
fungiert, angepaßt werden. Als Kamm kann man z.B. ein verhältnismäßig dünnes Stück
Plastik benutzen. Aus diesem müßten dann Teile herausgeschnitten werden, damit sich im
zu gießenden Gel Taschen für die aufzutrennende DNA bilden können.
Anstelle von Kohlefasergewebe können entweder Graphitminen aus Bleistiften oder
sogenannter V2A-Edelstahl, wie er in Kochtöpfen verwendet wird, eingesetzt werden. Der
Stromfluß ist bei allen drei Elektroden gleich (Tab. 18). Aluminiumfolie ist aufgrund
einer sehr schnell einsetzenden Elektrolyse nicht zu empfehlen. Neben Batterien als
Spannungsquelle kann auch auf einen gleichstromliefernden Spielzeugeisenbahntransformator
zurückgegriffen werden (Abb. 8). Allerdings sollte bei dessen Verwendung die angelegte
Spannung und die Stromstärke vom Lehrer überprüft werden. Um Gefährdungen durch
elektrische Schläge zu vermeiden, sollte die angelegte Spannung immer unter 25V liegen.
Eine Anleitung zum Bau einer Elektrophoreseeinheit ist dem Anhang (Kapitel 11.1)
beigefügt. Es sollte durchaus überlegt werden, ob Schüler z.B. im Kunstunterricht oder
in entsprechenden Arbeitsgemeinschaften am Bau beteiligt werden können. Bei
BAYRHUBER/ LUCIUS (1992) wird der Bau einer ähnlichen Einheit zur Elektrophorese
beschrieben. Dort wird die Verwendung eines Kunstoffdeckels einer Diaschachtel empfohlen,
der zumindest nach eigenen Erfahrungen nicht leicht zu beschaffen ist, da solche
deckeltragenden Diaboxen nur in Verbindung mit der Rahmung von Diapositiven im Fotolabor
vergeben werden.
Damit DNA im Gel sichtbar wird, muß diese angefärbt werden. Normalerweise verwendet
man dafür Ethidiumbromid, welches zwischen die Basenpaare der DNA interkaliert und bei
Bestrahlung mit UV-Licht orangefarbig fluoresziert. Beim Schülerexperiment wurde für die
Färbung der DNA am Ende der Elektrophorese bewußt auf die Verwendung von Ethidiumbromid
verzichtet. Der Verzicht erklärt sich aus mehreren Gründen: Erstens ist Ethidiumbromid
eine Substanz, die mutagen sowie stark giftig ist, zweitens muß Abfall, der diese
Substanz enthält, als Sondermüll entsorgt werden und drittens wird für die
Sichtbarmachung der so gefärbten DNA eine UV-Lampe benötigt, die nur in Ausnahmefällen
in Schulen vorhanden sein dürfte. Die Dokumentation der mit Ethidiumbromid angefärbten
DNA-Banden kann prinzipiell nur fotografisch erfolgen. Der dadurch erforderliche Einsatz
von Kamera, Entwicklung etc. ist von Schulen kaum zu leisten. Methylenblau stellt eine
Alternative zu Ethidiumbromid dar (NEVERS o.J.). Die mit Methylenblau angefärbte DNA
erscheint als blaue Bande im Gel und ist gut zu erkennen (Abb. 6, S. 110).
Die für Ethidiumbromid beschriebenen Nachteile treffen auf Methylenblau nicht zu. Zwar
ist Methylenblau in der Gefahrstoffliste (BAGUV 1995, Anlage 3, S. 40) beim Verschlucken
als gesundheitsschädlich ausgewiesen, allerdings erst ab einem Massenanteil in einer
Verdünnung von £ 25%. Da die Schüler jedoch nur mit
Verdünnungen von 1 bzw. 0,025% arbeiten, ist von keiner Gefährdung auszugehen. Entsorgt
werden kann Methylenblau in den verwendeten Konzentrationen in ausreichender Verdünnung
über den Ausguß. Beim Färbevorgang selbst sollte man mit Einweghandschuhen arbeiten, da
die Haut den Farbstoff vorübergehend annimmt und dieser nur relativ schwer zu entfernen
ist. Die Auswertung der Gele kann mit bloßem Auge erfolgen. Erleichtert wird die
Auswertung allerdings unter Weißlichtdurchleuchtung, wie sie z.B. mit Hilfe eines
Tageslichtprojektors geschehen kann. Als störend kann sich die höhere Nachweisgrenze
für DNA erweisen. Bei Methylenblau gefärbten Gelen liegt die Nachweisgrenze für DNA in
etwa bei 40 ng pro Bande und ist somit etwa viermal höher als bei einer Färbung mit
Ethidiumbromid.
Dennoch überwiegen die Vorteile von Methylenblau im Vergleich zu Ethidiumbromid. Deshalb
sollte man im Schülerexperiment immer auf Methylenblau zurückgreifen. Eine weitere
Alternative zu Ethidiumbromid besteht in dem im Lambda-Kit zur DNA-Färbung verwendeten
Azur A Farbstoff (Abb. 9). Die DNA-Färbelösung ist nach Angaben im Lehrerheft des
Lambda-Kits (MADDEN/ LUCIUS 1994) gesundheitsschädlich beim Verschlucken und reizt bei
Kontakt Augen und Haut. Durch geeigente Vorsichtmaßnahmen, wie z.B. das Tragen von
Handschuhen bei Handhabung und Hinweise des Lehrers, kann eine Gefährdung der Schüler
auf ein Minimum begrenzt werden. Entsorgt werden kann die DNA-Färbelösung in einem
Sammelgefäß für halogenierte organische Stoffe.
[Inhaltsverzeichnis] [Vorher] [Anfang] [Nächste]